
-
中文 | EN

中文 | EN
发布时间:2024-03-19 00:00:00.0
近日,刘聪课题组与同济大学生命科学与技术学院张勇课题组合作在Nature Communications杂志上发表了题为“Machine Learning Predictor Pspire Screens for Phase-Separating Proteins Lacking Intrinsically Disordered Regions”的论文。该研究成功开发了一种新的机器学习方法PSPire,用于预测不含有固有无序序列(intrinsically disordered regions,IDR)蛋白质的相分离。

蛋白质相分离与多种生理和病理过程紧密相关,通过生物信息学工具评估蛋白质的相分离能力对于推动相关科学领域的发展具有重要意义。尽管目前已经存在一些工具能够基于蛋白质序列信息预测含IDR蛋白的相分离,但这些工具在预测不含IDR蛋白的相分离时效果不佳。蛋白质相分离的驱动机制主要包括IDR之间的弱相互作用以及串联重复结构域之间的相互作用。因此,开发出能够准确评估不含IDR蛋白相分离能力的新工具显得尤为迫切。
在该研究中,作者首先利用AlphaFold预测蛋白质的结构,将这些结构与蛋白质的序列信息相结合,作为输入数据。随后,基于这些序列和结构特征,作者使用XGBoost分类器进行训练,成功开发了PSPire方法(图1)。PSPire能够有效地整合蛋白质的序列和结构特征,从而高效筛选出不含IDR的相分离蛋白质。在多个数据集上的评估结果表明,PSPire在预测不含IDR蛋白质相分离方面显示出了较现有工具更高的准确性。
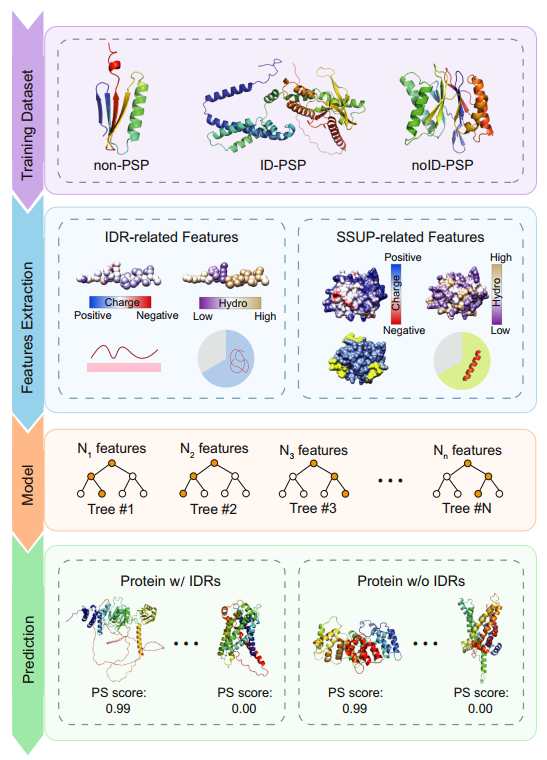
图1. PSPire 的工作流程。
进一步,作者通过体外相分离实验验证了PSPire预测结果的准确性。实验结果显示,几个不含IDR的相分离候选蛋白在细胞中形成了明显的凝聚体,与PSPire的预测结果一致。而预测为不发生相分离的阴性候选蛋白也符合预期,未观察到相分离现象(图2)。在测试的11个候选相分离蛋白中,有9个被证实能够发生相分离,这进一步验证了PSPire预测模型的有效性。综上,作者成功开发了用于预测不含IDR蛋白相分离的工具,弥补了现有预测工具的缺陷,为蛋白质相分离领域的研究提供了重要支持。
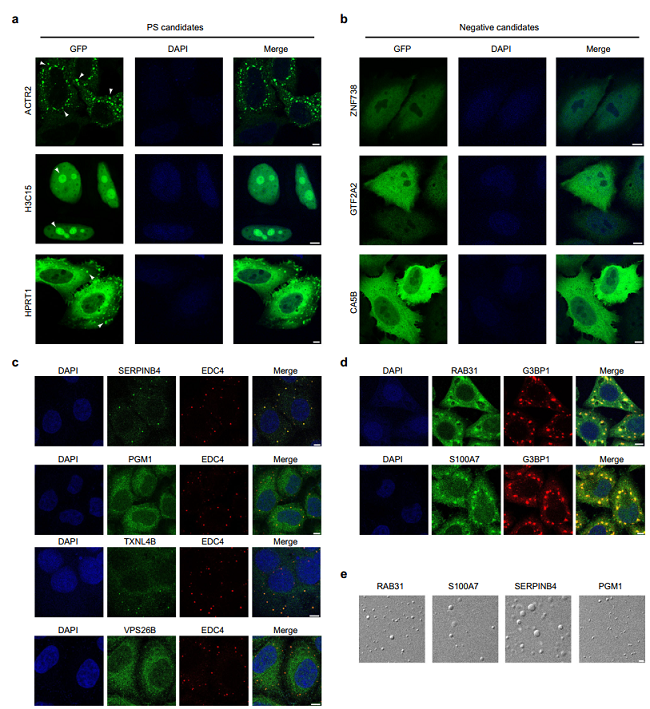
图2. PSPire 预测结果的体外验证。
刘聪研究员和同济大学张勇教授为该论文的共同通讯作者,张勇课题组博士研究生侯爽和刘聪课题组博士研究生胡姣姣为共同第一作者。该工作得到了国家自然科学基金委、国家科技部、上海市科委的支持。
原文链接:https://doi.org/10.1038/s41467-024-46445-y
