
-
中文 | EN

中文 | EN
发布时间:2025-07-29 00:00:00.0
硒蛋白(Selenoprotein)是一类含有硒代半胱氨酸(Selenocysteine, Sec, U)的特殊蛋白质。硒代半胱氨酸常被称为第21种氨基酸,是半胱氨酸(Cysteine, Cys, C)的类似物。硒蛋白因其独特的化学性质,在维持细胞氧化还原稳态和调控多种关键生理过程方面发挥着重要作用,并与许多疾病有关,包括神经退行性疾病、癌症、心血管疾病和糖尿病等。因此,深入解析硒蛋白的生物学特性对于阐明这些疾病背后的分子机制至关重要。然而,受限于Sec的稀有性以及这类蛋白或含Sec肽段的检测分析难度,目前人类仅鉴定出25种硒蛋白,全面绘制硒蛋白图谱及发现新的硒蛋白仍是重大挑战。
中国科学院上海有机化学研究所生物与化学交叉研究中心张耀阳课题组在前期研究中,开发了Sec特异性的质谱方法(Sec-specific mass spectrometry, SecMS)和非SECIS依赖的硒蛋白数据库(SECIS-independent selenoprotein database, SIS)(PMID: 30174312),首次在小鼠中绘制了组织特异性的硒蛋白图谱,并发现了多个新的硒蛋白,该成果为系统性研究硒蛋白质组提供了重要的理论和数据基础。近日,张耀阳课题组在 Advanced Science 上发表了一篇题为“DeepSecMS Advances DIA-Based Selenoproteome Profiling Through Cys-to-Sec Proxy Training”的研究论文。在该项研究中,研究人员基于深度学习和大规模代理数据训练,开发了DeepSecMS方法,深度解析了哺乳动物硒蛋白质组。

数据非依赖性采集(Data-independent acquisition, DIA)质谱方法近年来因其能够实现全面的数据采集以及准确且可重复的定量分析而备受关注。然而,传统的DIA分析通常需要数据依赖性采集(Data-dependent acquisition, DDA)实验生成的谱图库,但这种方法耗时且覆盖不完整。近年来,深度学习的发展使得预测谱图库成为可能,为常规及修饰肽段提供了一种无需DDA、准确且全面的替代方案。这种基于预测的策略在鉴定从未通过DDA分析捕获的新型硒蛋白方面尤具潜力。然而,由于已知的含硒肽段的谱图数量有限,基于深度学习的谱图预测难以直接应用于硒蛋白,严重限制了模型训练的准确性。
为解决这一科学问题,研究团队创新性地开发了DeepSecMS方法(图1),该方法基于Sec与Cys的化学相似性,研究人员采用了Cys-to-Sec取代训练策略,利用大量含Cys肽段数据,构建了大规模的理论Sec肽段的谱图库。结果表明,DeepSecMS能够精准预测Sec肽段的关键特征,包括MS2谱图、保留时间(RT)和离子淌度(IM)。将DeepSecMS与DIA技术相结合,在多种人源细胞系和小鼠组织样本中显著提升了已知硒蛋白的鉴定能力,更重要的是,还发现了大量高可信度的潜在的新的硒蛋白。这些成果充分展示了DeepSecMS在推动硒蛋白研究中的强大潜力。(图2)
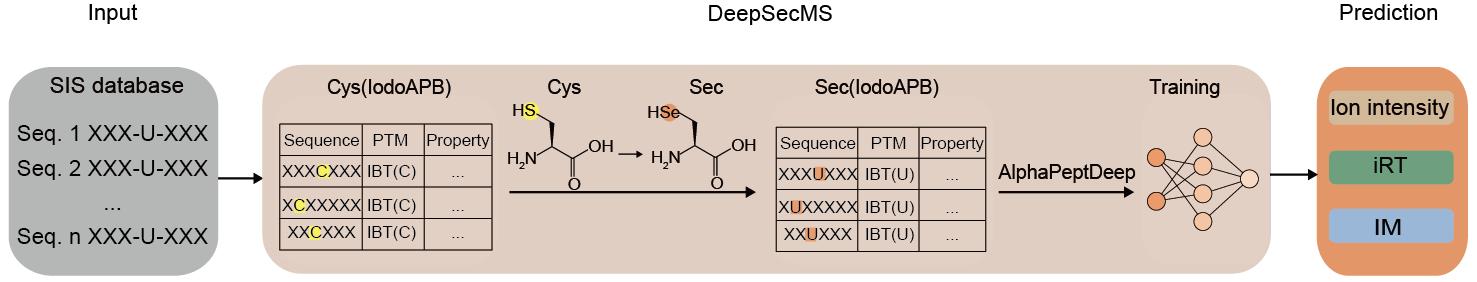
图1. DeepSecMS方法的实验流程。
综上,此项研究新开发的DeepSecMS方法,是一种鉴定硒蛋白的强大工具,并具有发现新型硒蛋白的巨大潜力。这一创新方法为硒蛋白组学研究带来了重要突破,提供了探索硒蛋白及其在人体健康与疾病中作用的新工具。更为重要的是,这一取代训练策略还可推广至其他低丰度蛋白变体或翻译后修饰的研究中,为推动微量蛋白质组学研究提供了具有可扩展性的技术框架。
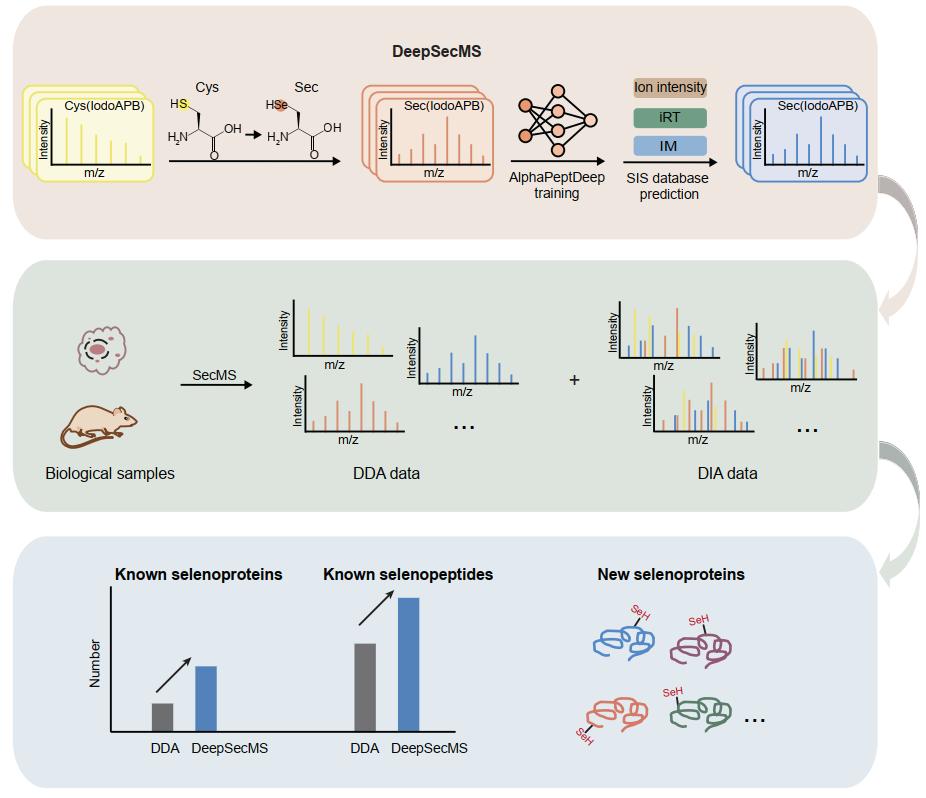
图2. 利用DeepSecMS深度解析哺乳动物硒蛋白质组。
中国科学院生物与化学交叉研究中心张耀阳研究员为本文的通讯作者,司晨芳博士为本文的第一作者。西湖大学的曾文锋研究员为该工作提供了重要的技术支持,复旦大学乔亮教授为该工作提供了宝贵的帮助。本项目得到了国家自然科学基金、中国科学院及上海市科委的基金支持。
原文链接:http://doi.org/10.1002/advs.202504109
